Projekte
Mediator probe PCR Design Software (MP PCR Desire)
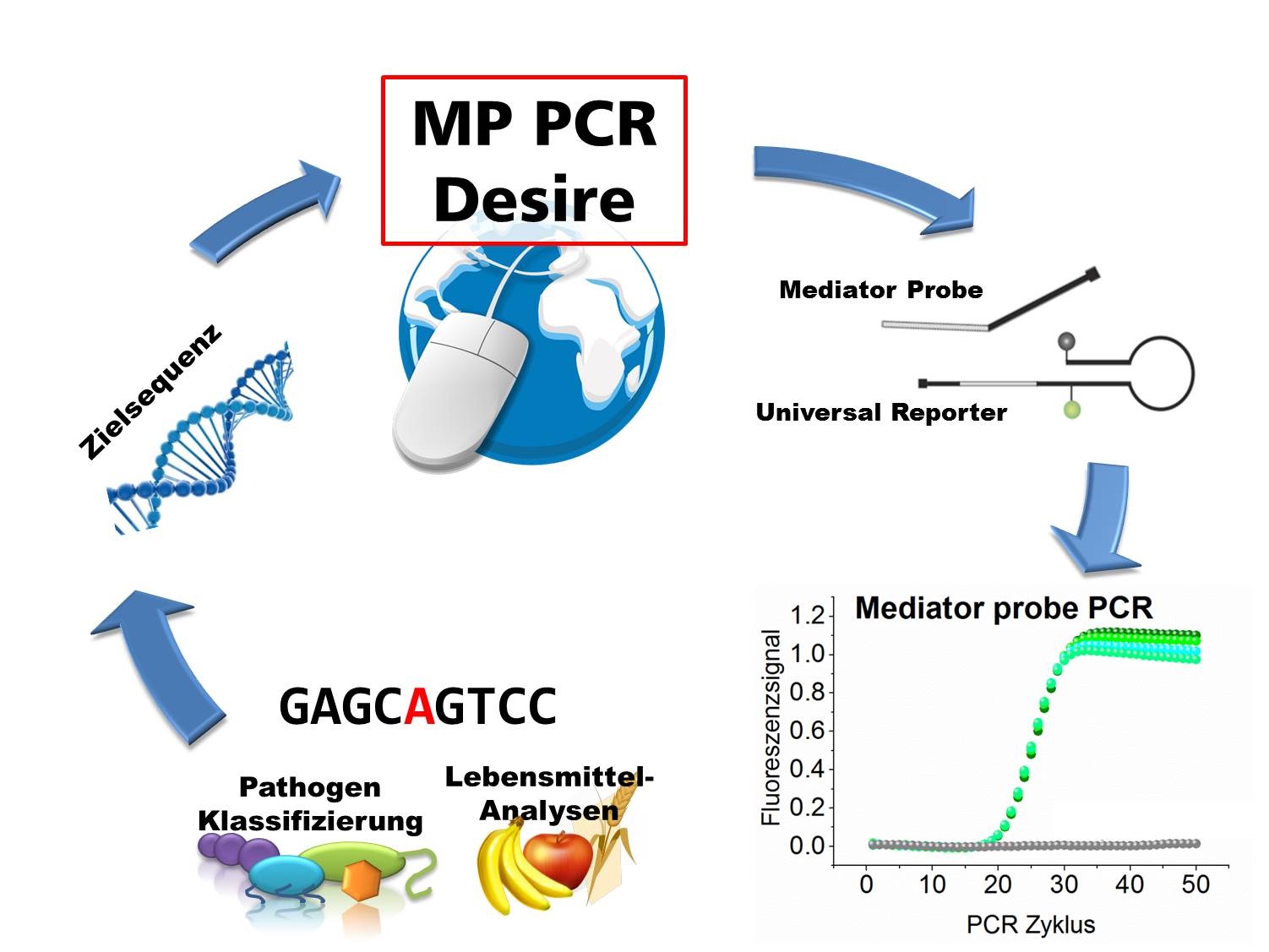
Projektbeschreibung
Die Mediatorsonden-PCR (engl.: Mediator Probe Assay) stellt eine Weiterentwicklung der Hydrolysesonden basierten real time PCR dar und zeichnet sich durch einen hochsignifikanten Signalanstieg, eine hohe Flexibilität beim Assay Design sowie der Verwendung kostengünstiger sequenzspezifischer DNA-Sonden aus. Dies wird durch eine Trennung von Detektion und Signalgenerierung erreicht. Hierbei dienen markierungsfreie Mediatorsonden (engl. mediator probe) der Detektion von DNA-Zielsequenzen. Diese werden in Gegenwart der DNA-Zielsequenz gespalten, wodurch eine Mediatorsequenz freigesetzt wird. Diese wiederum generiert an einem universellen Nachweismolekül (engl. universal reporter) ein Fluoreszenzsignal. Da Universal Reporter Zielsequenz unabhängig sind können sie für eine Großzahl unterschiedlicher PCR-Systeme eingesetzt werden. Weiterhin wird das von ihnen generierte Fluoreszenzsignal, im Gegensatz zu herkömmlichen Sondensystemen (z.B. Teqman-Sonden oder Molecular Beacons), nicht durch Sequenzcharakteristika beeinflusst. Am IMTEK konnten bereits über 30 Hydrolysesonden Systeme erfolgreich auf die Mediatorsonden-PCR übertragen werden, wobei die jeweilige Performance gleich oder besser der der Hydrolysesonden-PCR ist. Um die Mediatorsonden-PCR einem breiteren Spektrum an Anwendern zugänglich zu machen wir im Rahmen des Projektes MP-PCR-Desire eine Software entwickelt werden, welche auf Basis von vom Nutzer vorgegebene DNA-Zielsequenzen und Primern automatisiert Mediatorsequenzen und Universal Reporter ableitet.
Laufzeit
01.06.2015 bis 31.05.2017
Projektleitung
Dr. Michael Lehnert (Prof. Dr. Roland Zengerle)
Ansprechpartner/in
Dr. Michael Lehnert
Telefon:0761/203-73229
Kooperationspartner
Biomers.net
Universität Marburg, AG Rensing
Cap3 GmbH
Finanzierung
Zentrales Innovationsprogramm Mittelstand (ZIM)
Schlagworte
Bioanalytik, Biosensoren

